Mise à l'échelle des calculs SHAP avec PySpark et les UDF Pandas

Motivation
Avec la prolifération des applications des modèles de Machine Learning (ML) et surtout d'Apprentissage Profond (DL) dans la prise de décision, il devient de plus en plus crucial de percer le mystère de la boîte noire et de justifier les décisions commerciales clés basées sur les résultats de ces modèles. Par exemple, si un modèle de ML rejette la demande de prêt d'un client ou attribue un risque de crédit à un client donné dans le cadre de prêts entre particuliers, fournir aux parties prenantes une explication sur les raisons de cette décision pourrait être un outil puissant pour encourager l'adaptation des modèles. Dans de nombreux cas, le ML interprétable n'est pas seulement une exigence commerciale, mais aussi une exigence réglementaire pour comprendre pourquoi une certaine décision ou option a été proposée à un client. SHapley Additive exPlanations (SHAP) est un outil important que l'on peut exploiter pour l'IA explicable et pour aider à établir la confiance dans les résultats des modèles de ML et des réseaux de neurones pour la résolution de problèmes commerciaux.
SHAP est un framework de pointe pour l'explication de modèles, basé sur la théorie des jeux. L'approche consiste à trouver une relation linéaire entre les caractéristiques d'un modèle et la sortie du modèle pour chaque points de données de votre dataset. En utilisant ce cadre, vous pouvez interpréter la sortie de votre modèle de manière globale ou locale. L'interprétabilité globale vous aide à comprendre dans quelle mesure chaque caractéristique contribue aux résultats, de manière positive ou négative. D'autre part, l'interprétabilité locale vous aide à comprendre l'effet de chaque caractéristique pour une observation donnée.
Les implémentations SHAP les plus courantes, largement adoptées dans la Communauté de la Data Science, s'exécutent sur des machines à nœud unique, ce qui signifie qu'elles effectuent tous les calculs sur un seul cœur, quel que soit le nombre de cœurs disponibles. Par conséquent, elles ne tirent pas parti des capacités de calcul distribué et sont limitées par les contraintes d'un seul cœur.
Dans cet article, nous allons présenter un moyen simple de paralléliser les calculs de valeurs SHAP sur plusieurs machines, en particulier pour l'interprétabilité locale. Nous expliquerons ensuite comment cette solution monte en charge avec le nombre croissant de lignes et de colonnes dans le dataset. Enfin, nous mettrons en évidence certaines de nos conclusions sur ce qui fonctionne et ce qu'il faut éviter lors de la parallélisation des calculs SHAP avec Spark.
SHAP mononœud
Pour réaliser l'explicabilité, SHAP transforme un modèle en un Explainer ; les prédictions individuelles du modèle sont ensuite expliquées en leur appliquant l'Explainer. Il existe plusieurs implémentations des calculs de valeurs SHAP dans différents langages de programmation, dont une populaire en Python. Avec cette implémentation, pour obtenir des explications pour chaque observation, vous pouvez appliquer un explainer adapté à votre modèle. L'extrait de code suivant illustre comment appliquer un TreeExplainer à un classifieur Random Forest.
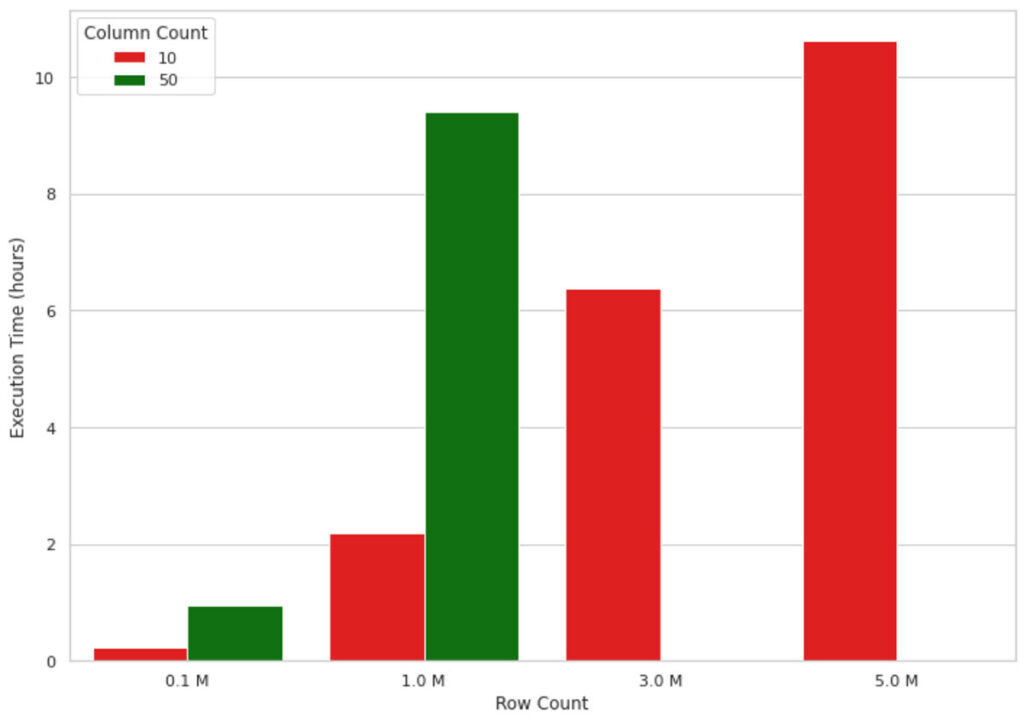
Cette méthode fonctionne bien pour de faibles volumes de données, mais lorsqu'il s'agit d'expliquer les résultats d'un modèle de ML pour des millions d'enregistrements, elle ne monte pas bien en charge en raison de la nature mononœud de l'implémentation. Par exemple, la visualisation de la figure 1 ci-dessous montre l'augmentation du temps d'exécution d'un calcul de valeur SHAP sur une machine mononœud (4 cœurs et 30,5 Go de mémoire) pour un nombre croissant d'enregistrements. La machine a manqué de mémoire pour des formes de données dépassant 1 million de lignes et 50 colonnes, par conséquent, ces valeurs sont manquantes sur la figure. Comme vous pouvez le constater, le temps d'exécution augmente de manière quasi linéaire avec le nombre d'enregistrements, ce qui n'est pas viable dans des scénarios réels. Attendre, par exemple, 10 heures pour comprendre pourquoi un modèle de machine learning a fait une prédiction n'est ni efficace ni acceptable dans de nombreux contextes professionnels.
Une façon de résoudre ce problème est d'utiliser le calcul approximatif. Vous pouvez définir l'argument approximate sur True dans la méthode shap_values. De cette façon, les divisions inférieures de l'arbre auront des poids plus élevés et il n'y a aucune garantie que les valeurs SHAP soient cohérentes avec le calcul exact. Cela accélérera les calculs, mais vous risquez d'obtenir une explication inexacte de la sortie de votre modèle. De plus, l'argument approximate n'est disponible que dans les TreeExplainers.
Une autre approche consisterait à tirer parti d'un framework de traitement distribué tel qu'Apache Spark™ pour paralléliser l'application de l'Explainer sur plusieurs cœurs.
Mise à l'échelle des calculs SHAP avec PySpark
Pour distribuer les calculs SHAP, nous travaillons avec cette implémentation Python et les UDF Pandas dans PySpark. Nous utilisons le dataset kddcup99 pour construire un détecteur d'intrusion réseau, un modèle prédictif capable de distinguer les mauvaises connexions, appelées intrusions ou attaques, des bonnes connexions normales. Ce dataset est connu pour présenter des défauts pour la détection d'intrusions. Cependant, dans cet article, nous nous concentrons uniquement sur les calculs de valeurs SHAP et non sur la sémantique du modèle de ML sous-jacent.
Les deux modèles que nous avons créés pour nos experimentations sont de simples classifieurs Random Forest entraînés sur des datasets avec 10 et 50 caractéristiques pour démontrer la scalabilité des solutions sur différentes tailles de colonnes. Veuillez noter que le dataset d'origine contient moins de 50 colonnes et que nous avons reproduit certaines de ces colonnes pour atteindre le volume de données souhaité. Les volumes de données avec lesquels nous avons expérimenté vont de 4 Mo à 1,85 Go.
Avant de nous plonger dans le code, donnons un aperçu rapide du fonctionnement des Dataframes Spark et des UDF. Les Dataframes Spark sont distribués (par lignes) sur un cluster, chaque groupe de lignes est appelé une partition et chaque partition (par défaut) peut être traitée par 1 cœur. C'est ainsi que Spark réalise fondamentalement le traitement parallèle. Les UDF Pandas sont un choix naturel, car pandas peut facilement alimenter SHAP et est performant. Un UDF pandas, parfois appelé UDF vectorisé, offre de meilleures performances que les UDF Python en utilisant Apache Arrow pour optimiser le transfert de données.
L'extrait de code ci-dessous montre comment paralléliser l'application d'un Explainer avec une UDF Pandas dans PySpark. Nous définissons une UDF pandas appelée calculate_shap, puis nous passons cette fonction à mapInPandas. Cette méthode est ensuite utilisée pour appliquer la méthode parallélisée au dataframe PySpark. Nous utiliserons cette UDF pour exécuter nos tests de performance SHAP.
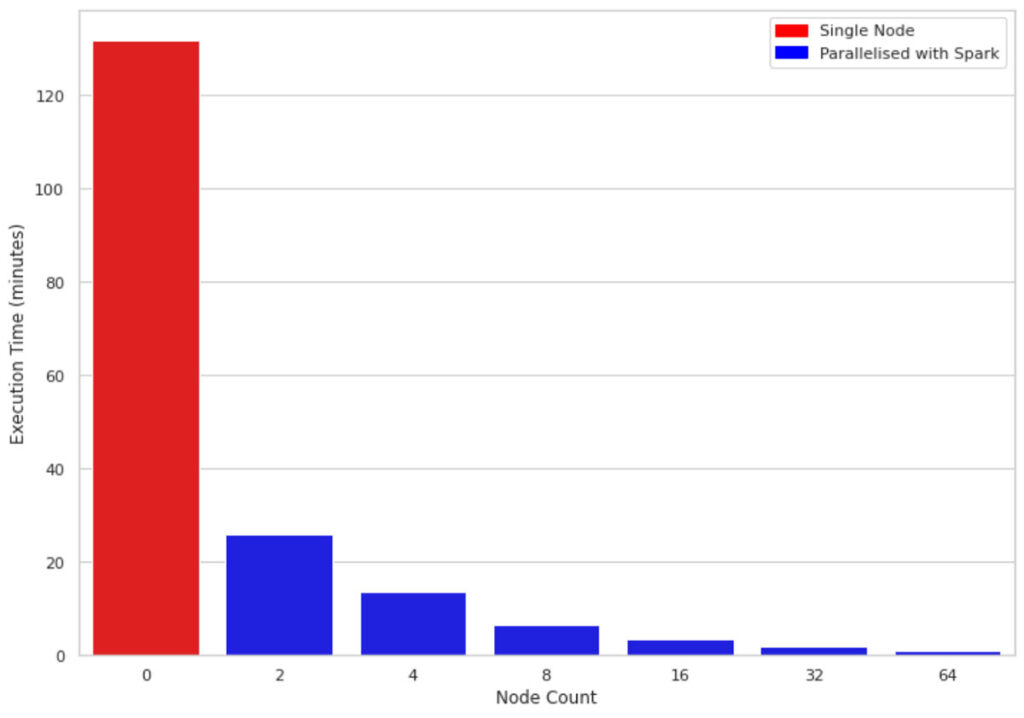
La figure 2 compare le temps d'exécution pour 1 million de lignes et 10 colonnes sur une machine à nœud unique par rapport à des clusters de tailles 2, 4, 8, 16, 32 et 64 respectivement. Les machines sous-jacentes pour tous les clusters sont similaires (4 cœurs et 30,5 Go de mémoire). Une observation intéressante est que le code parallélisé tire parti de tous les cœurs sur les nœuds du cluster. Par conséquent, même l'utilisation d'un cluster de taille 2 améliore les performances de près de 5 fois.
Mise à l'échelle avec l'augmentation du volume de données
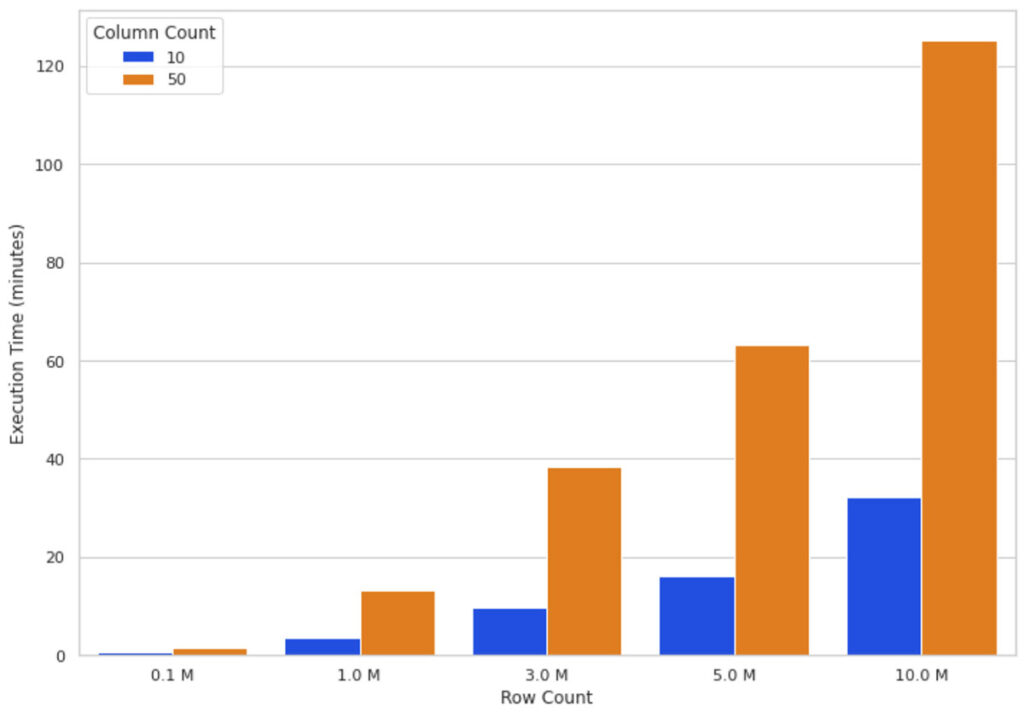
En raison de la manière dont SHAP est implémenté, les caractéristiques supplémentaires ont un impact plus important sur les performances que les lignes supplémentaires. Nous savons maintenant que les valeurs SHAP peuvent être calculées plus rapidement en utilisant Spark et les UDF Pandas. Ensuite, nous examinerons comment SHAP se comporte avec des caractéristiques/colonnes supplémentaires.
Intuitivement, l'augmentation de la taille des données signifie que l'algorithme SHAP doit effectuer plus de calculs. La figure 3 illustre les temps d'exécution des valeurs SHAP sur un cluster de 16 nœuds pour différents nombres de lignes et de colonnes. Vous pouvez voir que la mise à l'échelle des lignes augmente le temps d'exécution de manière presque directement proportionnelle, c'est-à-dire que doubler le nombre de lignes double presque le temps d'exécution. L'augmentation du nombre de colonnes a une relation proportionnelle avec le temps d'exécution ; l'ajout d'une colonne augmente le temps d'exécution de près de 80 %.
Ces observations (Figure 2 et Figure 3) nous ont amenés à conclure que plus vous avez de données, plus vous pouvez monter en charge votre calcul horizontalement (en ajoutant plus de nœuds de travail) pour maintenir un temps d'exécution raisonnable.
Quand envisager la parallélisation ?
Les questions auxquelles nous voulions répondre sont les suivantes : quand la parallélisation en vaut-elle la peine ? Quand devrait-on commencer à utiliser PySpark pour paralléliser les calculs SHAP, même en sachant que cela pourrait alourdir le calcul ? Nous avons mis en place une Experimentation pour mesurer l'effet du doublement de la taille du cluster sur l'amélioration du temps d'exécution des calculs SHAP. L'objectif de l'Experimentation est de déterminer quelle taille de données justifie l'ajout de plus de Ressources horizontales (c'est-à-dire l'ajout de plus de nœuds worker) au problème.
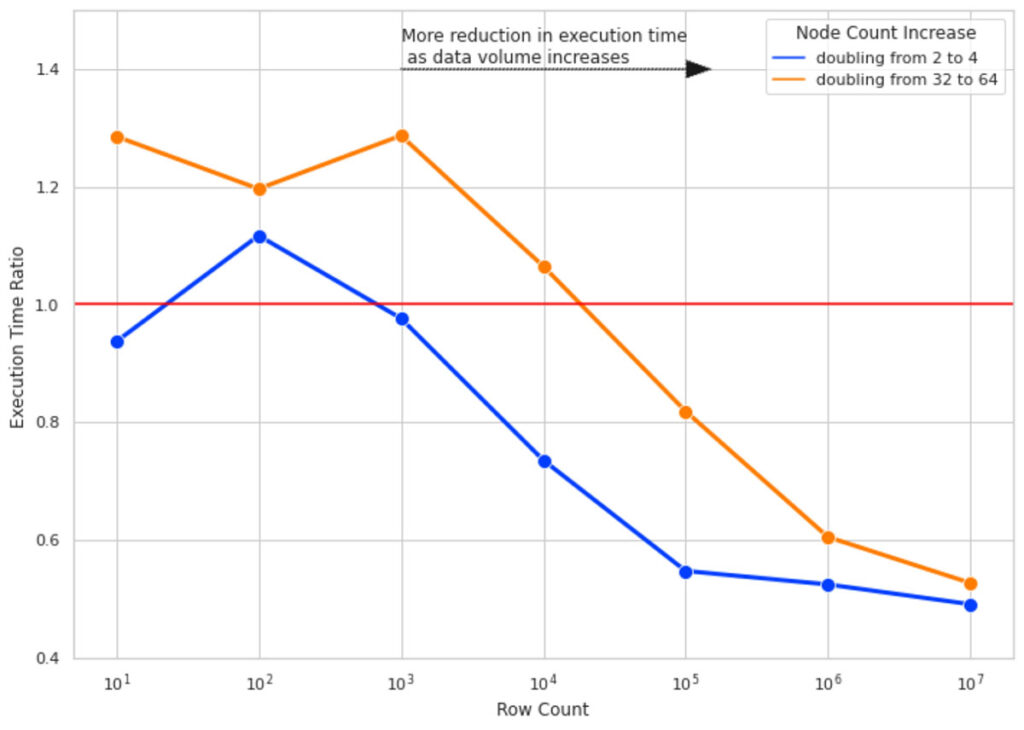
Nous avons exécuté les calculs SHAP pour 10 colonnes de données et pour des nombres de lignes de 10, 100, 1000, etc. jusqu'à 10 millions. Pour chaque nombre de lignes, nous avons mesuré le temps d'exécution du calcul SHAP 4 fois pour des tailles de cluster de 2, 4, 32 et 64. Le ratio de temps d'exécution est le rapport entre le temps d'exécution du calcul de la valeur SHAP sur les plus grandes tailles de cluster (4 et 64) et l'exécution du même calcul sur une taille de cluster avec la moitié du nombre de nœuds (respectivement 2 et 32).
La figure 4 illustre le résultat de cette Experimentation. Voici les points clés à retenir :
-
- Pour un faible nombre de lignes, le doublement de la taille des clusters n'améliore pas le temps d'exécution et, dans certains cas, l'aggrave en raison de la surcharge ajoutée par la gestion des tâches Spark (d'où un ratio de temps d'exécution > 1).
- À mesure que nous augmentons le nombre de lignes, doubler la taille du cluster devient plus efficace. Pour 10 millions de lignes de données, doubler la taille du cluster réduit presque de moitié le temps d'exécution.
- Quel que soit le nombre de lignes, doubler la taille du cluster de 2 à 4 est plus efficace que de la doubler de 32 à 64 (remarquez l'écart entre les lignes bleue et orange). À mesure que la taille de votre cluster augmente, le surcoût lié à l'ajout de nœuds supplémentaires augmente également. Ce phénomène s'explique par des tailles de partition pour lesquelles la taille des données par partition est trop faible, car il est plus coûteux de créer une tâche distincte pour traiter cette petite quantité de données que d'utiliser une taille de données/partition plus optimale.
Pièges
Repartitionnement
Comme mentionné ci-dessus, Spark met en œuvre le parallélisme par le biais de la notion de partitions ; les données sont partitionnées en blocs de lignes et chaque partition est traitée par un seul cœur par défaut. Lorsque les données sont initialement lues par Apache Spark, il se peut qu'il ne crée pas nécessairement de partitions optimales pour le calcul que vous souhaitez exécuter sur votre cluster. En particulier, pour le calcul des valeurs SHAP, nous pouvons potentiellement obtenir de meilleures performances en repartitionnant notre dataset.
Il est important de trouver un équilibre entre créer des partitions suffisamment petites et ne pas les rendre si petites que le surcoût de leur création l'emporte sur les avantages de la parallélisation des calculs.
Pour notre test de performance, nous avons décidé d'utiliser tous les cœurs du cluster à l'aide du code suivant :
Pour des volumes de données encore plus importants, vous pouvez définir le nombre de partitions à 2 ou 3 fois le nombre de cœurs. La clé est d'expérimenter et de trouver la meilleure stratégie de partitionnement pour vos données.
Utilisation de display()
Si vous travaillez sur un Notebook Databricks, vous voudrez peut-être éviter d'utiliser la fonction display() lors de l'évaluation des temps d'exécution. L'utilisation de display() peut ne pas nécessairement vous montrer la durée d'une Transformations complète ; elle a une limite de lignes implicite, qui est injectée dans la query et, selon l'Opérations que vous voulez mesurer, par exemple, l'écriture dans un fichier, il y a une surcharge supplémentaire pour rassembler les résultats vers le driver. Nos temps d'exécution ont été mesurés à l'aide de la méthode d'écriture de Spark en utilisant le format « noop ».
Conclusion
Dans ce billet de blog, nous avons présenté une solution pour accélérer les calculs SHAP en les parallélisant avec PySpark et les UDF Pandas. Nous avons ensuite évalué les performances de la solution sur des volumes de données croissants, différents types de machines et des configurations changeantes. Voici les principaux points à retenir :
-
-
- Le calcul SHAP sur un seul nœud augmente de façon linéaire avec le nombre de lignes et de colonnes.
- La parallélisation des calculs SHAP avec PySpark améliore les performances en exécutant les calculs sur tous les CPU de votre cluster.
- L'augmentation de la taille du cluster est plus efficace lorsque vous avez des volumes de Big Data plus importants. Pour les petits jeux de données, cette méthode n'est pas efficace.
-
Travaux futurs
Mise à l'échelle verticale - L'objectif de cet billet de blog était de montrer comment la mise à l'échelle horizontale avec de grands datasets peut améliorer les performances du calcul des valeurs SHAP. Nous avons commencé du principe que chaque nœud de notre cluster disposait de 4 cœurs et de 30,5 Go. À l'avenir, il serait intéressant de tester les performances de la mise à l'échelle verticale et horizontale ; par exemple, en comparant les performances entre un cluster de 4 nœuds (4 cœurs, 30,5 Go chacun) et un cluster de 2 nœuds (8 cœurs, 61 Go chacun).
Sérialisation/Désérialisation - Comme mentionné, l'une des raisons principales d'utiliser les UDF Pandas plutôt que les UDF Python est que les UDF Pandas utilisent Apache Arrow pour améliorer la sérialisation/désérialisation des données entre la JVM et le processus Python. Il pourrait y avoir des optimisations potentielles lors de la conversion des partitions de données Spark en batchs d'enregistrements Arrow, et expérimenter avec la taille de batch Arrow pourrait entraîner des gains de performance supplémentaires.
Comparaison avec les implémentations SHAP distribuées - Il serait intéressant de comparer les résultats de notre solution aux implémentations distribuées de SHAP, telles que Shparkley. En menant une telle étude comparative, il serait important de s'assurer en premier lieu que les résultats des deux solutions sont comparables.
(Cet article de blog a été traduit à l'aide d'outils basés sur l'intelligence artificielle) Article original
Ne manquez jamais un article Databricks
Et ensuite ?

Data Science e ML
October 31, 2023/9 min de leitura
